Marre de jouer à Angry Birds ou à Candy Crush ? Les généticiens de Norwich (Royaume-Uni) ont pensé à vous et viennent de lancer un nouveau jeu sur Facebook, le réseau social numéro 1 dans le monde. Baptisé Fraxinus, ce jeu vous enrôle dans une aventure scientifique au cœur du génome du frêne commun (Fraxinus excelsior) et de l’un de ses parasites, le champignon Chalara fraxinea, responsable de la maladie du flétrissement du frêne, également appelée chalarose. qui pourrait entraîner sa quasi disparition du continent européen. Ainsi, fin 2012, soit seulement quatre ans après l’identification d’un premier foyer en Haute-Saône, 39 départements français étaient déjà touchés par cette épiphytie.
Le projet OpenAshDieBack, regroupant plusieurs laboratoires (le laboratoire Sainsbury, le Centre John Innes et le Centre d’analyse génomique) tous basés à Norwich, au nord de Londres, a été lancé en décembre 2012 alors que les séquençages des génomes du frêne et du champignon responsable de la chalarose (identifié en 2006) venaient d’être achevés. Face à cet afflux de données génétiques, les biologistes anglais ont immédiatement décidé de les rendre publiques, avant même d’avoir eu le temps de les analyser eux-mêmes. Il faut dire que la tâche s’avère immense : le génome de Chalara fraxinea comporte 63 millions de paires de bases, et celui du frêne 954 millions ! Les chercheurs du monde entier réfléchissent ainsi aux moyens de faire face à ce “déluge” de données issues du séquençage. Dans un récent article publié dans la revue Genome Biology, trois biologistes américains proposent de s’inspirer de l’astronomie, qui a dû elle aussi gérer un “tsunami” de données, en créant un cyberécosystème dédié à cette tâche, et appellent de leur vœu l’émergence d’une “astrogénomique” s’appuyant sur des standards de fichiers communs et des bases de données en libre accès, comme a pu le faire la NASA.
Les cyber-joueurs appelés à l’aide
En attendant l’avènement d’une NASA de la génétique, les chercheurs se sont appuyés, comme les astronomes avant eux, sur une autre source : le crowdsourcing. Pionnier dans le domaine, le projet SETI@home, lancé en 1999, utilise les capacités de calcul des ordinateurs de millions de volontaires pour analyser les ondes captées par un radio-télescope à la recherche de traces d’une intelligence extra-terrestre. Depuis, de nombreux travaux de biologie ont vu le jour en enrôlant des participants en dehors des laboratoires de recherche : c’est le cas par exemple de FoldIt, où les joueurs doivent replier une protéine pour déterminer la structure tridimensionnelle qui minimise son énergie (une tâche complexe qui nécessite une très importante puissance de calcul). Leur contribution essentielle a été reconnue par la communauté scientifique, et les “FoldIt players” figurent ainsi parmi la liste des auteurs d’un article publié en 2010 dans la prestigieuse revue Nature. Depuis, ces programmes de sciences citoyennes ou participatives se sont multipliés (pour en citer quelques-uns : Phylo, EteRNA, EyeWire, BioGames ou encore DarwinTunes, évoqué dans un précédent article), jusqu’à adopter les supports web les plus répandus, comme le réseau social Facebook où a pris pied Fraxinus.
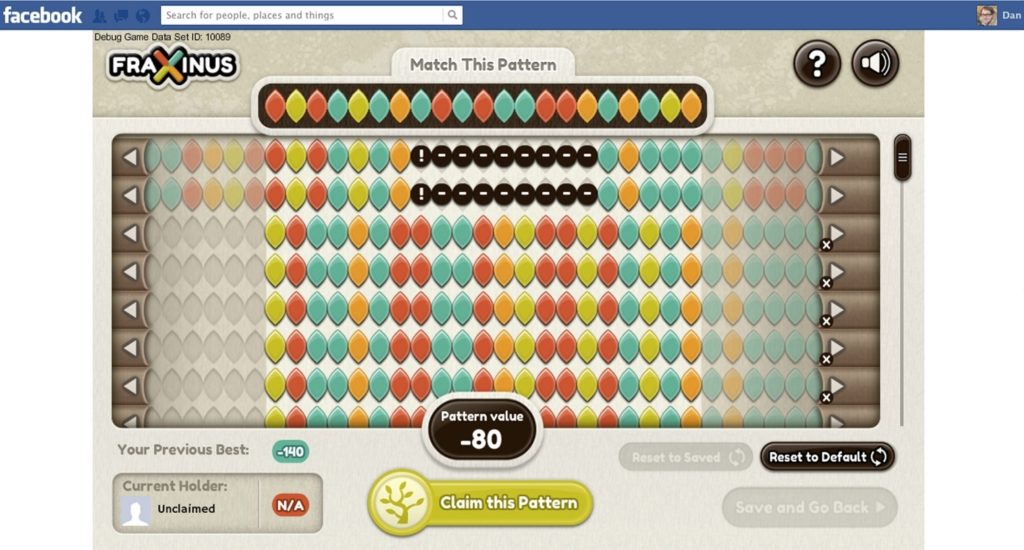
Le séquençage génomique produit une multitude de fragments d’ADN, ne contenant que quelques dizaines de paires de base, ce qui complique la reconstitution de la séquence complète de l’ADN (un problème évoqué dans un précédent article au sujet de l’ADN de la Peste Noire). Le but de Fraxinus n’est pas de “recoller” les fragments d’ADN issus du séquençage des génomes du frêne et du champignon parasite, mais de comparer les séquences obtenues chez plusieurs spécimens de chacune de ces espèces afin de déceler d’éventuelles différences. Parmi ces mutations génétiques présentes de façon sporadique, certaines pourraient être associées à une plus grande pathogénicité de souches de champignons ou à la résistance de certains arbres face à l’infection.
Cette tâche est habituellement confiée à un ordinateur, mais elle s’avère longue et compliquée, d’où l’idée de la déléguer à des participants volontaires dont les facultés d’observation et d’analyse dépassent celles des systèmes informatiques. Le jeu repose sur des séquences de 21 bases, représentées par quatre couleurs symbolisant les quatre bases du code génétique (adénine, guanine, cytosine et thymine). Le but est d’aligner au mieux deux séquences voisines obtenues chez deux spécimens distincts en introduisant un minimum de variations, par l’insertion ou la délétion de bases. Le joueur n’a besoin d’aucune connaissance en génétique : il n’a qu’à bouger les briques colorées pour faire coïncider au mieux les deux séquences proposées par le jeu, afin d’obtenir le score le plus haut parmi les autres joueurs en réduisant au maximum l’écart entre ces deux séquences colorées. Pour Dan MacLean, l’un des concepteurs de Fraxinus, “le principal défi est de combler le fossé entre les joueurs et les chercheurs : si le jeu n’est pas intéressant pour le joueur, le projet va échouer, quel que soit l’intérêt scientifique pour le chercheur“. À vous de jouer, et de juger si la mission est remplie.
Source : D. MacLean, Cutting edge: changing the rules of the game, eLife, 13 août 2013.
Crédit photo : Jessica – Wikimedia Commons (CC BY 2.0).





