Qui n’a pas rêvé de réaliser dans sa cuisine la célèbre recette du Pudding à l’arsenic immortalisée par le dessin animé Astérix et Cléopâtre, bien entendu à des fins scientifiques afin de vérifier si ce gâteau fait réellement mal au ventre (comme la réaction d’Obélix permet d’en douter) ? Encore faut-il pour cela retrouver le venin de cobra, le vitriol ou bien entendu l’arsenic utilisé par Amonbofis et son scribe Tournevis. C’est sur ces bases que fonctionne la science : pour valider un résultat, il faut pouvoir le reproduire, ce qui implique de communiquer de façon claire sur les conditions dans lesquelles l’expérience première a été réalisée pour qu’elle puisse être répétée par une autre équipe. Dans une étude publiée par la revue PeerJ, Nicole Vasilevsky, de l’université de l’Oregon (États-Unis), et ses collègues se sont demandé si les chercheurs (en biologie) respectaient tous cette règle de base de la recherche. Les résultats sont pour le moins… décevants.
La question de la reproductibilité d’une expérience scientifique est centrale : elle a ainsi permis de démonter la théorie de la “mémoire de l’eau” avancée par Jacques Benveniste en 1988. En effet, la revue Nature avait diligenté une enquête contradictoire qui avait échoué à réitérer les expériences montrant que l’eau pourrait garder en “mémoire” les propriétés de composés chimiques ou biologiques qu’elle avait côtoyés par le passé (une théorie à la base de l’homéopathie…). Plus récemment, une étude de la compagnie pharmaceutique Amgen a montré que seuls 11 % des travaux académiques publiés avaient pu être reproduits dans leurs laboratoires. Comment expliquer ce chiffre alarmant ? En se penchant en détail sur la section « Materials and Methods », qui figure dans tout article de science expérimentale : celle-ci décrit le matériel utilisé ainsi que les protocoles mis au point pour obtenir résultats nouveaux et conclusions inédites obtenus par l’équipe rédactrice. C’est la tâche à laquelle s’est attelé l’équipe de Nicole Vasilevsky, qui a épluché 238 publications issues de 84 revues de sciences de la vie pour vérifier si les matériels utilisés étaient décrits avec suffisamment de précision pour permettre à d’autres de reproduire l’expérience de façon indépendante.
Retrouver un anticorps dans une botte de foin…
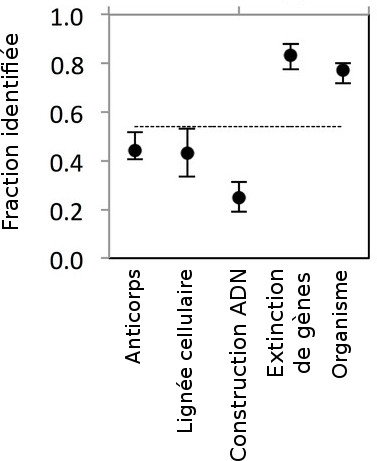
Parmi les 1 700 “objets” biologiques utilisés dans les articles répertoriés, les chercheurs n’ont pu en identifier de façon certaine qu’un peu plus de la moitié (54 %). La proportion de matériels identifiables à partir des informations fournies par les auteurs des publications varie selon leur nature. Au rayon des bons élèves, les organismes (souris, mouches, levures ou poissons zèbre) et les réactifs permettant l’extinction de gènes. Par ce terme, on désigne les morpholinos ou les ARN interférents qui bloquent l’expression d’un gène et donc la synthèse de la protéine associée (à ce sujet, je vous recommande l’excellent article de Pierre Barthélémy sur les mécanismes épigénétiques sur lesquels repose la prochaine génération d’OGM). Parmi les matériels plus difficilement identifiables par le lecteur attentif d’une publication scientifique, on retrouve les anticorps, souvent utilisés pour reconnaître une molécule d’intérêt dans une cellule (seuls un quart des anticorps sont identifiés par leur référence de catalogue, pourtant disponible auprès des fournisseurs), les lignées cellulaires (dont il est de toute façon difficile de garantir l’origine en raison de nombreuses contaminations au cours de leur exploitation) et surtout les constructions ADN, dont seules un quart sont identifiables de façon certaine, par leur séquence intégrale.
L’analyse de ces résultats montre que les revues les plus prestigieuses (distinguées par un fort facteur d’impact) sont également touchées par cette difficulté à identifier les matériels utilisés par les auteurs des publications. Ce sont même celles qui ont les consignes à ce sujet les moins contraignantes (les articles publiés dans Nature ou Science sont courts, et la section Materials et Methods en fait souvent les frais). Si certaines revues imposent des normes plus strictes, comme le Journal of Comparative Neurology, ces règles ne sont pas toujours contrôlées de façon énergique par les reviewers qui évaluent l’article pour publication. Pour Nicole Vasilevsky et ses collègues, « le manque de prise de conscience et les difficultés à coordonner l’action des parties prenantes sont à l’origine du problème ».
Les auteurs de cette étude, qui cherchent autant à observer la situation actuelle de la recherche en sciences du vivant qu’à interpeller les acteurs du domaine, insistent : “Nous sommes convaincus qu’à moins que les chercheurs puissent accéder aux matériels de recherche spécifiques utilisés dans les études publiées, ils continueront à reproduire avec difficulté les découvertes de leurs pairs et à les prolonger par d’autres travaux.” En effet, si la publicité des données et des codes est une étape cruciale (comme l’illustrent le site RunMyCode ou Thomas Piketty, qui rend publiques toutes les données utilisées pour son dernier livre, Le capital au XXIe siècle) pour reproduire l’analyse des résultats ou de les utiliser pour d’autres travaux, il est tout aussi important de s’assurer que les données rendues publiques puissent elles aussi être reproduites. Il faut donc soutenir les diverses initiatives qui permettent d’avancer dans ce domaine en facilitant l’identification des matériels biologiques, parmi lesquelles BioSharing, eagle-i ou Antibody Registry qui proposent des systèmes d’identifications universels pour faciliter la communication entre laboratoires. Car la confiance dans la recherche et ses découvertes dépend avant tout de son ouverture et de sa transparence, gage de sa falsificabilité chère à Karl Popper.
Source : N.A. Vasilevsky, On the reproducibility of science: unique identification of research resources in the biomedical literature, PeerJ, 5 septembre 2013.
Crédit photo : Amarand Agasi – Flickr (CC BY-NC-SA 2.0).





